## install new packages IN THE CONSOLE!
install.packages("pacman")6 Einlesen von Daten
Importieren von Datendateien
Lernziele
In diesem Kapitel werden wir lernen, wie man:
- lokale Datendateien mit dem Paket
readrzu importieren - mit fehlenden Werten umzugehen
- Variablen in Faktoren umwandeln
Lesungen
Die Pflichtlektüre zur Vorbereitung auf dieses Thema ist Kap. 8 (Data Import) in Wickham et al. (2023).
Eine ergänzende Lektüre ist Ch. 4 (Data Import) in Nordmann & DeBruine (2022).
Wiederholung
Im letzten Kapitel haben wir das Gelernte in die Praxis umgesetzt. Wir haben einen Datensatz aus dem languageR-Paket (Baayen & Shafaei-Bajestan, 2019) eingelesen, ihn mit dem dplyr-Paket aus dem tidyverse verarbeitet und mehrere Diagramme mit dem ggplot2-Paket aus dem tidyverse erstellt. All dies wurde mit einem Quarto-Skript durchgeführt.
6.1 Einrichtung
6.1.1 Pakete mit pacman
Zu Beginn werden wir mit dem Paket pacman beginnen. Die Funktion p_load() nimmt Paketnamen als Argumente und prüft dann, ob Sie das Paket installiert haben. Wenn ja, dann lädt sie das Paket (genau wie library()). Wenn Sie das Paket nicht installiert haben, wird das Paket installiert und geladen (wie mit install.packages(), library()). Das erspart uns, neue Pakete einzeln zu installieren, und bedeutet auch, dass, wenn wir unser Skript mit anderen teilen, sie einfach pacman::p_load() ausführen können.
## load packages
pacman::p_load(tidyverse, ## wrangling
janitor, ## wrangling
here ## relative file paths
)Wir haben nun tidyverse geladen, und die neuen Pakete janitor und here installiert und geladen. Um mehr über diese Pakete herauszufinden, versuchen Sie ?janitor und ?here in der Konsole einzugeben.
6.1.2 RProjects
Bevor wir mit unserer ersten Datei beginnen, müssen wir sicherstellen, dass wir innerhalb unseres RProjekts arbeiten. Zu Beginn des Kurses haben Sie eine ZIP-Datei von GitHub (https://github.com/daniela-palleschi/r4ling_student) heruntergeladen, die einige Ordner und eine .RProj-Datei enthielt. Hoffentlich haben Sie bis jetzt innerhalb dieses RProjekts gearbeitet und Ihre Skripte im Ordner notizen gespeichert. Von nun an wird es notwendig sein, innerhalb dieses RProjekts zu arbeiten, damit wir immer auf unsere relevanten Datendateien zugreifen können, die in einem Ordner namens daten gespeichert werden sollten.
Um ein RProjekt zu öffnen, navigieren Sie einfach zu dem Ordner auf Ihrem Rechner und doppelklicken Sie auf die .RProj-Datei. Wenn Sie sich bereits in RStudio befinden, können Sie auch überprüfen, ob Sie im richtigen RProjekt arbeiten, indem Sie oben im Fenster nachsehen.
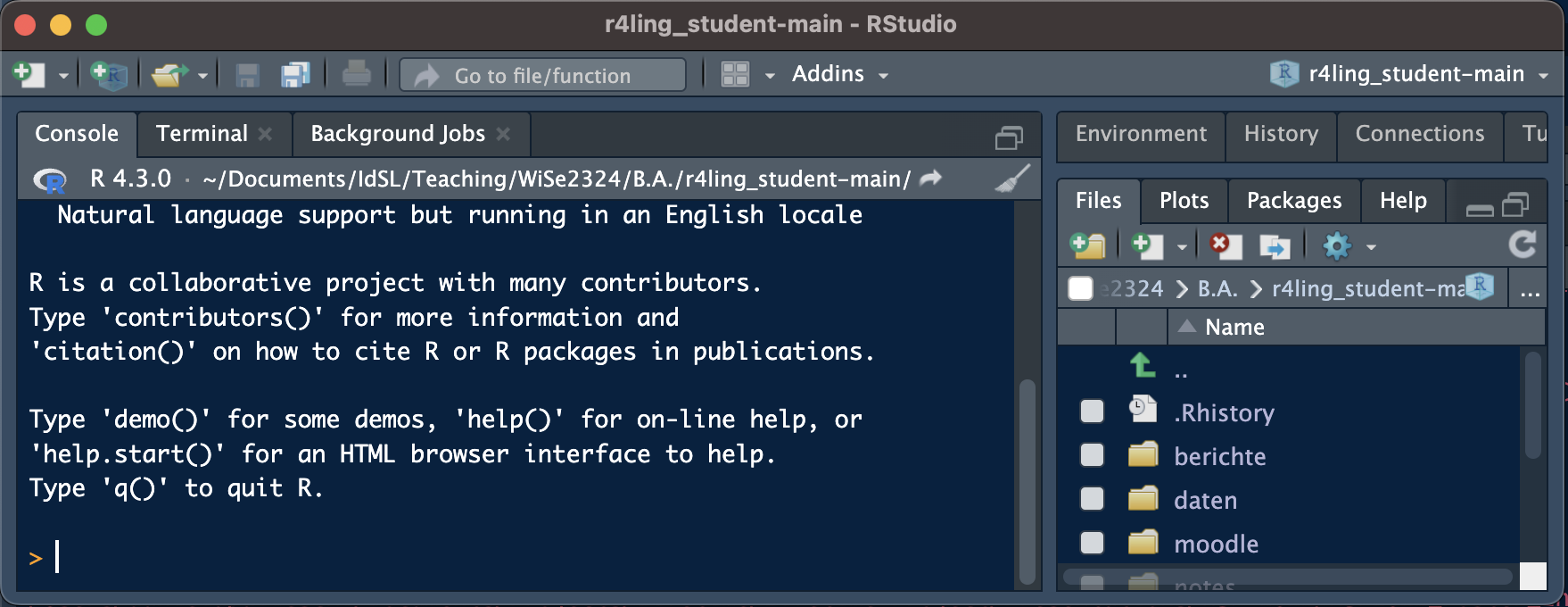
- Überprüfen Sie, ob Sie tatsächlich in Ihrem RProjekt arbeiten. Wenn dies der Fall ist, sehen Sie
r4ling_student-mainoben in Ihrem RStudio (Abbildung 6.1).
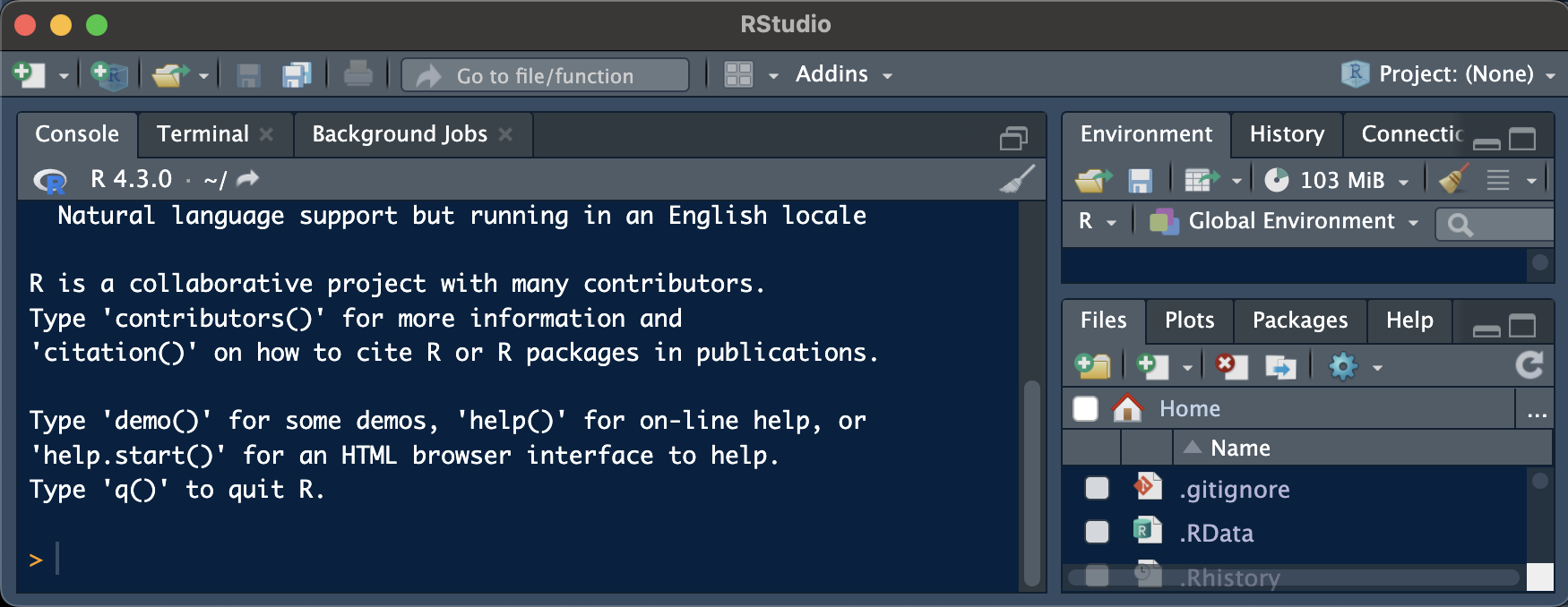
- Wenn dies nicht der Fall ist (Abbildung 6.2), können Sie zum RProjekt wechseln, indem Sie auf die Schaltfläche “Projekt” oben rechts im RStudio klicken (Abbildung 6.3; Hinweis: Die Screenshots stammen von einem Mac, auf einem Windows-Rechner sieht es etwas anders aus).
- Fügen Sie Ihrem RProject-Ordner einen Ordner namens
datenhinzu.
6.2 CSV: Komma getrennter Wert
Bisher haben wir mit Daten aus dem R-Paket languageR gearbeitet. Daten aus Paketen sind eine großartige Möglichkeit, die Werkzeuge der Datenwissenschaft zu erlernen, aber normalerweise wollen wir mit unseren eigenen Daten arbeiten, nicht mit eingebauten Spielzeugdaten. Wir werden uns nur auf rechteckige Daten (d. h. aufgeräumte Daten) konzentrieren, obwohl Ihre Daten zu Beginn oft nicht aufgeräumt sind. Es gibt viele verschiedene Dateitypen, die Daten annehmen können, z. B. .xlsx, .txt, .csv, .tsv. Der Dateityp “csv” ist der häufigste Dateityp und steht für: Comma Separated Values.
So sieht eine einfache CSV-Datei aus:
Student ID,Full Name,favourite.food,mealPlan,AGE
1,Sunil Huffmann,Strawberry yoghurt,Lunch only,4
2,Barclay Lynn,French fries,Lunch only,5
3,Jayendra Lyne,N/A,Breakfast and lunch,7
4,Leon Rossini,Anchovies,Lunch only,
5,Chidiegwu Dunkel,Pizza,Breakfast and lunch,five
6,Güvenç Attila,Ice cream,Lunch only,6Die erste Zeile (die “Kopfzeile”) enthält die Spaltennamen. Die folgenden Zeilen enthalten die Daten. Wie viele Variablen gibt es? Wie viele Beobachtungen?
Wir lernen jetzt etwas über aufgeräumte Daten und sehen uns ein Beispiel an. Anschließend werden wir eine CSV-Datei in R laden.
Versuchen Sie, .xlsx-Dateien im Allgemeinen zu vermeiden, vor allem aber, wenn Sie Ihre Daten in R laden wollen. Der Grund dafür ist, dass Excel viele Formatierungsprobleme hat, die für R problematisch sind. Wenn Sie einen Excel-Datensatz haben, versuchen Sie, ihn als .csv zu speichern, bevor Sie ihn in R einlesen (Datei > Speichern unter > Dateiformat > Komma-getrennter Wert).
6.2.1 Tidydaten
Unabhängig davon, in welchem Format Ihre Daten vorliegen, sollten sie aufgeräumt sein. Das bedeutet erstens, dass die Daten rechteckig sein sollten und dass jede Spalte eine Variable, jede Zeile eine Beobachtung und jede Zelle einen Datenpunkt darstellt (Abbildung 6.4).

6.3 Tabelle zu csv
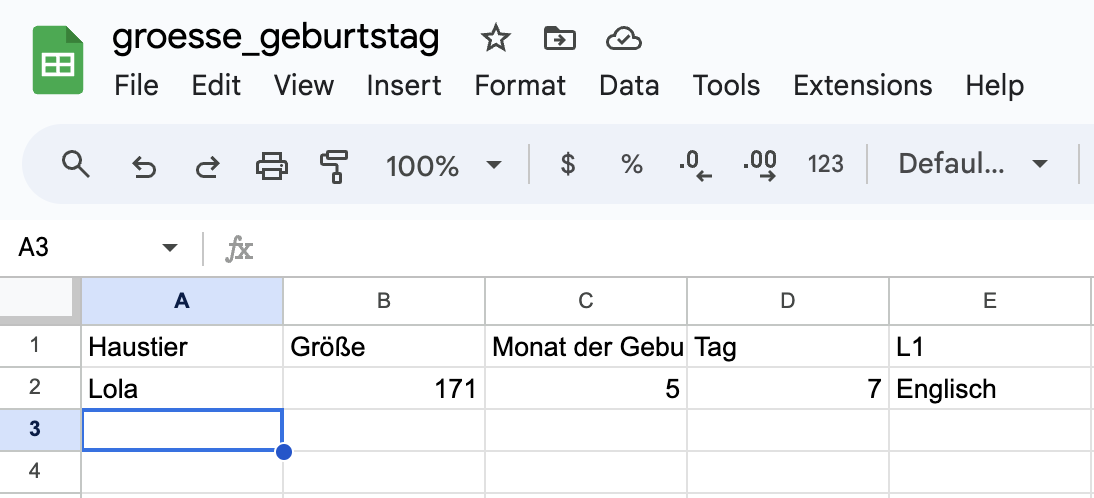
Lassen Sie uns einige Spielzeugdaten in einem Arbeitsblatt sammeln, das wir dann als CSV-Datei speichern und in R laden werden. Klicken Sie hier, um zu einem bearbeitbaren Arbeitsblatt zu gelangen. Geben Sie die relevanten Informationen über sich selbst ein, oder erfinden Sie einige Daten: den Namen eines Haustiers, das Sie haben/hatten, Größe, Geburtsmonat und -tag sowie Ihre erste Sprache. Wenn Sie kein Haustier haben, lassen Sie die Zelle leer.
6.3.1 CSV speichern
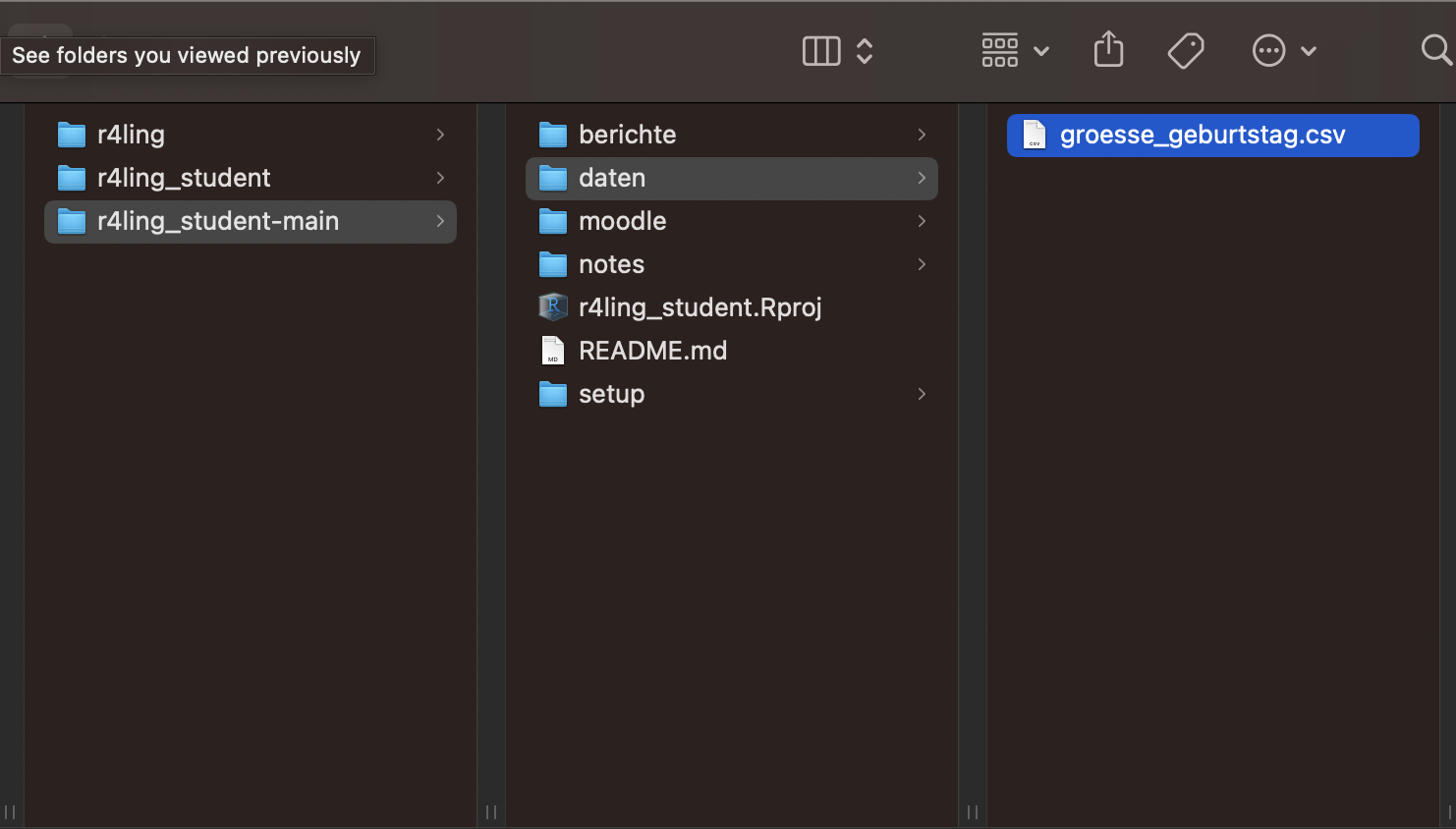
Jetzt müssen wir unser Arbeitsblatt als CSV-Datei auf unserem Computer speichern. Solange wir in unserem RProjekt arbeiten, wird R immer nach Dateien aus dem Ordner suchen, der unser RProjekt enthält. Stellen wir also zunächst sicher, dass unser Ordner einen Unterordner namens daten enthält. Darin werden wir alle unsere Daten speichern.
Speichern einer CSV
Beispiel 6.1
- Erstellen Sie einen neuen Ordner mit dem Namen
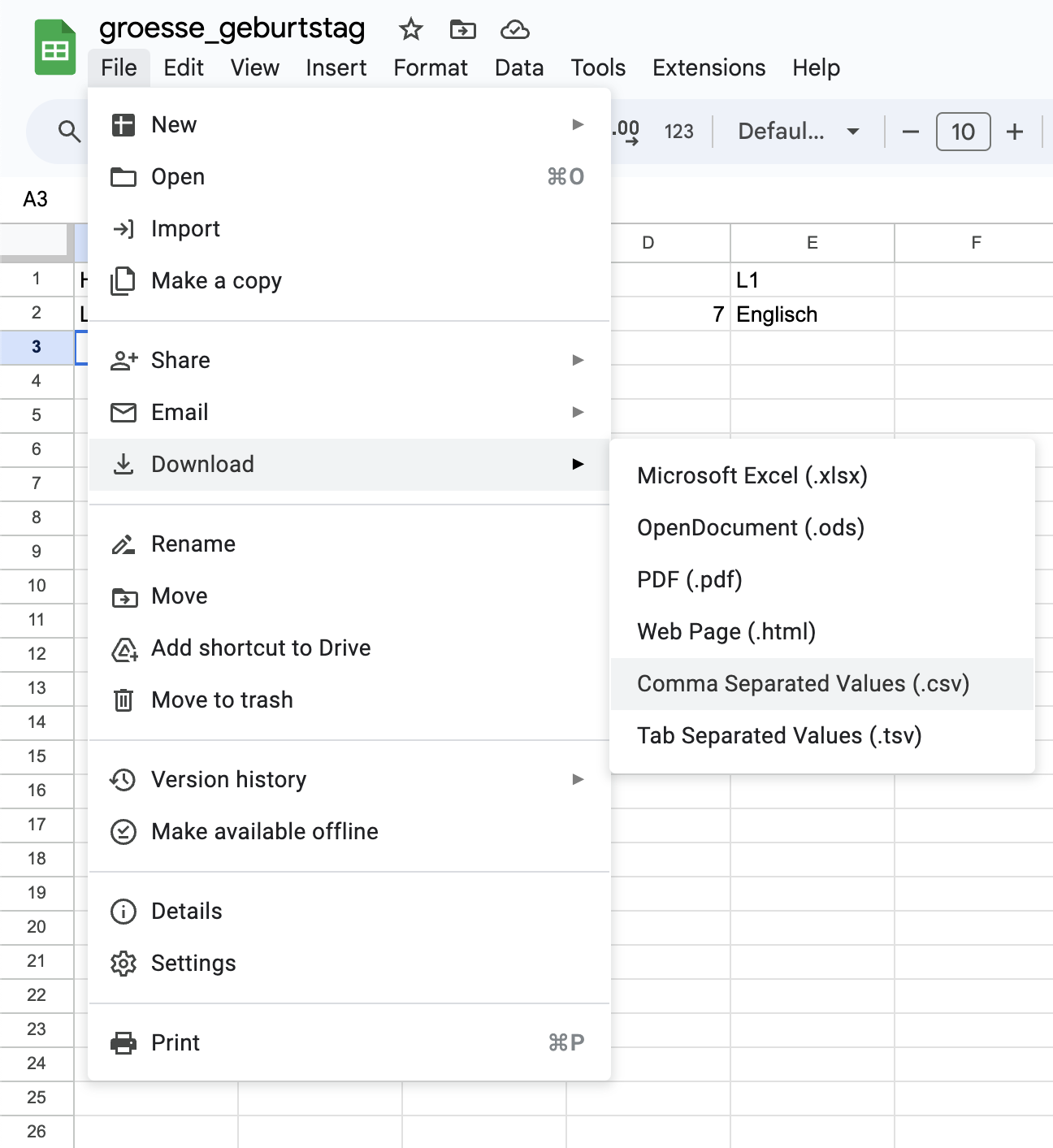
datenin Ihrem Projektordner (falls Sie das nicht schon getan haben). - Laden Sie das Google Sheet herunter und speichern Sie es in Ihrem
daten-Ordner alsgroesse_geburtstag.csv. - Gehen Sie zu Ihrem
daten-Ordner und überprüfen Sie, ob die CSV-Datei dort ist.
6.4 Das readr-Paket
Unsere Daten können als Tabelle angezeigt werden, genau wie unsere eingebauten Datensätze aus dem languageR-Paket (Baayen & Shafaei-Bajestan, 2019). Genau wie bei den eingebauten Datensätzen müssen wir zuerst die Daten einlesen, aber anstatt nur den Namen des eingebauten Datensatzes anzugeben, müssen wir eine Funktion verwenden, die CSV-Daten liest. Wir müssen auch angeben, wo sich die Daten in unserem RProject-Ordner befinden.
Das Paket readr (Teil von tidyverse) kann die meisten Datentypen einlesen und hat mehrere Funktionen für verschiedene Datentypen.
read_csv(here::here("daten", "groesse_geburtstag.csv"))| Größe | Geburtsmonat | L1 | Haustier | Was für ein Haustier? |
|---|---|---|---|---|
| 171 | 5 | Englisch | Lola | Hundin |
| 168 | 11 | Deutsch | keine | keine |
| 182 | 4 | Deutsch | N/A | NA |
| 190 | 8 | Deutsch | Knut | Kater |
| 170 | 10 | Deutsch | Emma | Hundin |
| 163 | 2 | Deutsch | Üzgür | Kater |
| 164 | 7 | Italienisch | Fipsy | Katze |
| 167 | 12 | Schwedisch | Anna | Fisch |
| 189 | 10 | Norwegisch | Arvid | Papagei |
| 163 | 7 | Russisch | Narzis | Kater |
| 159 | 11 | Punjabi | Mimi | Katze |
| 173 | 9 | Deutsch | Percy | Hund |
df_groesse
Beispiel 6.2
- Importieren Sie den Datensatz “groesse_geburtstag.csv” und speichern Sie ihn als Objekt mit dem Namen “df_groesse”.
df_ist die Abkürzung für DataFrame; es ist eine gute Idee, ein Präfix vor Objektnamen zu verwenden, damit wir wissen, was jedes Objekt enthält.
- Wenn Daten mit
read_csvimportiert werden, werden einige Informationen in der Konsole ausgegeben. Was wird gedruckt? - Untersuche den Datensatz mit Funktionen wie
summary()oderhead() - Sehen Sie etwas Ungewöhnliches?
6.5 Das here-Paket
Aber woher weiß R genau, wo der Ordner daten zu finden ist? Unser Arbeitsverzeichnis ist auf den Ort unseres RProjekts auf unserem Computer festgelegt. Wann immer wir auf Daten in unserem RProjekt zugreifen wollen, sollten wir here() verwenden (vorausgesetzt, wir haben das here-Paket bereits geladen). Um zu sehen, von wo aus here() startet, führen Sie here() aus. Wie sieht die Ausgabe im Vergleich zu der von getwd() (für ‘get working directory’)?
here()[1] "/Users/danielapalleschi/Documents/IdSL/Webbooks/r4ling"getwd()[1] "/Users/danielapalleschi/Documents/IdSL/Webbooks/r4ling/mats"Die Ausgabe wird auf allen unseren Rechnern anders aussehen, aber was gleich sein sollte, ist unsere Ordnerstruktur innerhalb unserer Projekte (z. B. data/groesse_geburtstag.csv).

here-Paket
Vor dem here-Paket mussten wir R explizit mitteilen, wo sich eine Datei auf unserem Computer befindet (z.B., /Users/danielapalleschi/Documents/IdSL/Teaching/SoSe23/BA/ba_daten/daten/students.csv), oder die Funktion setwd() (set Working Directory) benutzen, um R mitzuteilen, wo alle Dateien zu finden sind (z.B. setwd("/Users/danielapalleschi/Documents/IdSL/Teaching/SoSe23/BA/ba_daten")). Glücklicherweise brauchen Sie diese absoluten Dateipfade oder setwd() nie zu benutzen!
Aus der hier-Paketdokumentation:
The goal of the here package is to enable easy file referencing in project-oriented workflows. In contrast to using
setwd(), which is fragile and dependent on the way you organize your files, here uses the top-level directory of a project to easily build paths to files.
Das bedeutet, dass wir nun den großen Vorteil haben, dass wir unseren Projektordner überall hin verschieben können und unser Dateipfad immer noch relativ zu dem Ort ist, an den wir unseren Projektordner verschoben haben. Das bedeutet, dass das Projekt unabhängig davon läuft, wo es sich auf Ihrem Computer befindet. Sie können auch jemandem den Projektordner schicken, und alles sollte auf dessen Rechner laufen!
6.6 Arbeit mit Daten
Daten sind chaotisch, aber mit Erfahrung können wir lernen, sie zu bändigen. Im Folgenden finden Sie einige Tipps, die das Gelernte über die Datenverarbeitung erweitern.
6.6.1 Fehlende Werte
Bei der Datentransformation geht es darum, unsere Daten zu “reparieren”, wenn sie nicht “in Ordnung” sind. In unserem df_groesse Datenrahmen haben Sie vielleicht einige NA oder N/A Werte bemerkt. N/A” wurde in einer unserer Beobachtungen als Text geschrieben und wird von R als solcher gelesen. N/A” in R bezieht sich auf fehlende Daten (“Nicht verfügbar”). Echte fehlende Werte sind komplett leer, so dass N/A in unseren df_groesse-Daten nicht wirklich als fehlender Wert gelesen wird, obwohl wir möchten, dass R weiß, dass dies als fehlende Daten zählt und nicht das Haustier von jemandem namens “NA” (Menschen tun seltsame Dinge!). Um dies zu beheben, können wir das Argument na = für die Funktion read_csv() verwenden, das der Funktion read_csv() mitteilt, welche Werte sie mit fehlenden Werten gleichsetzen soll.
# force "N/A" to missing values
df_groesse <- read_csv(here::here("daten", "groesse_geburtstag.csv"),
na = "N/A")
# print the head of the data set
head(df_groesse)# A tibble: 6 × 5
Größe Geburtsmonat L1 Haustier `Was für ein Haustier?`
<dbl> <dbl> <chr> <chr> <chr>
1 171 5 Englisch Lola "Hundin"
2 168 11 Deutsch keine "keine"
3 182 4 Deutsch <NA> ""
4 190 8 Deutsch Knut "Kater"
5 170 10 Deutsch Emma "Hundin"
6 163 2 Deutsch Üzgür "Kater" Jetzt wird der Wert, der vorher "" war, als NA gelesen. Aber was ist mit der leeren Zelle? Wir haben jetzt überschrieben, dass read_csv() leere Zellen als NA liest. Jetzt wollen wir read_csv() anweisen, mehr als eine Art von Eingabe als NA zu lesen, d.h. wir wollen es anweisen, "" und "N/A" als NA zu lesen. Dafür verwenden wir unsere immer nützliche Verkettungsfunktion: c().
# force "N/A" and empty cells to missing values
df_groesse <- read_csv(here::here("daten", "groesse_geburtstag.csv"),
na = c("N/A",""))
# print the head of the data set
head(df_groesse)# A tibble: 6 × 5
Größe Geburtsmonat L1 Haustier `Was für ein Haustier?`
<dbl> <dbl> <chr> <chr> <chr>
1 171 5 Englisch Lola Hundin
2 168 11 Deutsch keine keine
3 182 4 Deutsch <NA> <NA>
4 190 8 Deutsch Knut Kater
5 170 10 Deutsch Emma Hundin
6 163 2 Deutsch Üzgür Kater 6.6.2 Spaltennamen
Wenn wir df_groesse in der Konsole ausdrucken, werden wir sehen, dass ein Spaltenname von Backticks umgeben ist (z.B. `Monat der Geburt). Das liegt daran, dass er ein Leerzeichen enthält, das syntaktisch nicht gültig ist (Variablennamen müssen mit einem Buchstaben beginnen und dürfen keine Leerzeichen oder Sonderzeichen enthalten). Eine schnelle Lösung ist die Funktion clean_names() aus dem Paket janitor, das wir bereits geladen haben.
clean_names(df_groesse)# A tibble: 12 × 5
grosse geburtsmonat l1 haustier was_fur_ein_haustier
<dbl> <dbl> <chr> <chr> <chr>
1 171 5 Englisch Lola Hundin
2 168 11 Deutsch keine keine
3 182 4 Deutsch <NA> <NA>
4 190 8 Deutsch Knut Kater
5 170 10 Deutsch Emma Hundin
6 163 2 Deutsch Üzgür Kater
7 164 7 Italienisch Fipsy Katze
8 167 12 Schwedisch Anna Fisch
9 189 10 Norwegisch Arvid Papagei
10 163 7 Russisch Narzis Kater
11 159 11 Punjabi Mimi Katze
12 173 9 Deutsch Percy Hund Das sieht besser aus! Aber wenn Sie jetzt head(df_groesse) ausführen, sehen Sie dann die bereinigten Spaltennamen?
Das sollten Sie nicht, denn wenn wir ein Objekt durch eine Funktion übergeben, wird das Objekt nicht ‘aktualisiert’, so dass wir das Objekt erneut mit dem Zuweisungsoperator <- zuweisen müssen.
df_groesse <- janitor::clean_names(df_groesse)Aber wir wissen oft, dass wir mehrere Funktionen (read_csv(), clean_names()) auf demselben Objekt ausführen wollen, denken Sie daran, dass wir das mit Pipes tun können.
6.6.3 Pipes
Pipes werden am Ende eines Funktionsaufrufs eingefügt, wenn das Ergebnis dieser Funktion durch eine nachfolgende Funktion weitergegeben werden soll. Pipes können als “und dann…” gelesen werden.
read_csv(here::here("daten", "groesse_geburtstag.csv")) |>
head()# A tibble: 6 × 5
Größe Geburtsmonat L1 Haustier `Was für ein Haustier?`
<dbl> <dbl> <chr> <chr> <chr>
1 171 5 Englisch Lola Hundin
2 168 11 Deutsch keine keine
3 182 4 Deutsch N/A <NA>
4 190 8 Deutsch Knut Kater
5 170 10 Deutsch Emma Hundin
6 163 2 Deutsch Üzgür Kater Derzeit gibt es 2 Pipes, die in R verwendet werden können.
- die
magrittrPaket-Pipe:%>% - die neue native R-Pipe:
|>
Es gibt keine großen Unterschiede, die für unsere aktuellen Anwendungen wichtig sind, also benutzen wir die neue |>. Sie können die Tastenkombination Cmd/Ctrl + Shift/Strg + M verwenden, um eine Pipe zu erzeugen. Dies könnte die “Magrittr”-Paket-Pipe erzeugen, was in Ordnung ist, aber wenn Sie das ändern möchten, können Sie das unter Werkzeuge > Globale Optionen > Code > Native Pipe-Operator verwenden tun.
Beispiel 6.3
- Laden Sie den Datensatz
groesse_geburtstag.csverneut mit festenNAs und dann- Benutze eine Pipe, um
clean_names()auf dem Datensatz aufzurufen, und dann - rufen Sie die Funktion “head()” auf
- Überprüfen Sie die Anzahl der Beobachtungen und Variablen, gibt es ein Problem?
- Benutze eine Pipe, um
- Laden Sie den Datensatz
groesse_geburtstag.csverneut mit festenNAs, speichern Sie ihn als Objektdf_groesse, und dann- Verwenden Sie eine Pipe, um
clean_names()auf den Datensatz anzuwenden.
- Verwenden Sie eine Pipe, um
- Warum sollte man nicht eine Pipe und die Funktion
head()verwenden, wenn man den Datensatz als Objekt speichert?
6.6.4 Variablentypen
Das Paket readr errät den Typ der Daten, die jede Spalte enthält. Die wichtigsten Spaltentypen, die man kennen muss, sind numerisch und Faktor (kategorisch). Faktoren enthalten Kategorien oder Gruppen von Daten, können aber manchmal aussehen wie numerische Daten. Zum Beispiel enthält unsere Spalte “Monat” Zahlen, aber sie könnte auch den Namen jedes Monats enthalten. Ein guter Weg, um zu wissen, was was ist: Es ist sinnvoll, den Mittelwert einer “numerischen” Variablen zu berechnen, aber nicht den eines “Faktors”. Zum Beispiel ist es sinnvoll, den Mittelwert der Körpergröße zu berechnen, aber nicht den Mittelwert des Geburtsmonats.
Um sicherzustellen, dass eine Variable als Faktor gespeichert wird, können wir die Funktion as_factor() verwenden. Wir können entweder die R-Basissyntax verwenden, um dies zu tun, indem wir ein $ verwenden, um eine Spalte in einem Datenrahmen zu indizieren:
df_groesse$geburtsmonat <- as_factor(df_groesse$geburtsmonat)Or we can use tidyverse syntax and the mutate() function.
df_groesse <-
df_groesse |>
mutate(geburtsmonat = as_factor(geburtsmonat))6.7 Andere Dateitypen und Begrenzungszeichen
Sobald Sie mit read_csv() vertraut sind, sind die anderen Funktionen von readr einfach zu benutzen, Sie müssen nur wissen, wann Sie welche benutzen.
Die Funktion read_csv2() liest Semikolon-separierte Dateien. Diese verwenden Semikolons (;) anstelle von Kommas (,), um Felder zu trennen und sind in Ländern üblich, die , als Dezimaltrennzeichen verwenden (wie Deutschland).
Die Funktion read_tsv() liest Tabulator-getrennte Dateien. Die Funktion read_delim() liest Dateien mit beliebigen Trennzeichen ein und versucht, das Trennzeichen zu erraten, es sei denn, Sie geben es mit dem Argument delim = an (z.B. read_delim(students.csv, delim = ",")).
readr hat mehrere andere Funktionen, die ich persönlich noch nicht gebraucht habe, wie zum Beispiel:
read_fwf()liest Dateien mit fester Breiteread_table()liest eine gängige Variante von Dateien mit fester Breite, bei der die Spalten durch Leerzeichen getrennt sindread_log()liest Log-Dateien im Apache-Stil
Weitere Übungen
Weitere Übungen zu diesem Kapitel finden Sie in Kapitel A.6.
Lernziele 🏁
Heute haben wir gelernt, wie man…
- lokale Datendateien mit dem Paket
readrimportiert ✅ - fehlende Werte behandeln ✅
- Variablen in Faktoren umwandeln ✅
Lassen Sie uns nun dieses neue Wissen anwenden.
Session Info
Hergestellt mit R version 4.4.0 (2024-04-24) (Puppy Cup) und RStudioversion 2023.9.0.463 (Desert Sunflower).
sessionInfo()R version 4.4.0 (2024-04-24)
Platform: aarch64-apple-darwin20
Running under: macOS Ventura 13.2.1
Matrix products: default
BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
locale:
[1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8
time zone: Europe/Berlin
tzcode source: internal
attached base packages:
[1] stats graphics grDevices datasets utils methods base
other attached packages:
[1] magick_2.8.3 here_1.0.1 janitor_2.2.0 lubridate_1.9.3
[5] forcats_1.0.0 stringr_1.5.1 dplyr_1.1.4 purrr_1.0.2
[9] readr_2.1.5 tidyr_1.3.1 tibble_3.2.1 ggplot2_3.5.1
[13] tidyverse_2.0.0
loaded via a namespace (and not attached):
[1] utf8_1.2.4 generics_0.1.3 renv_1.0.7 stringi_1.8.3
[5] hms_1.1.3 digest_0.6.35 magrittr_2.0.3 evaluate_0.23
[9] grid_4.4.0 timechange_0.3.0 fastmap_1.1.1 rprojroot_2.0.4
[13] jsonlite_1.8.8 fansi_1.0.6 scales_1.3.0 cli_3.6.2
[17] crayon_1.5.2 rlang_1.1.3 bit64_4.0.5 munsell_0.5.1
[21] withr_3.0.0 yaml_2.3.8 parallel_4.4.0 tools_4.4.0
[25] tzdb_0.4.0 colorspace_2.1-0 pacman_0.5.1 vctrs_0.6.5
[29] R6_2.5.1 lifecycle_1.0.4 snakecase_0.11.1 htmlwidgets_1.6.4
[33] bit_4.0.5 vroom_1.6.5 pkgconfig_2.0.3 pillar_1.9.0
[37] gtable_0.3.5 glue_1.7.0 Rcpp_1.0.12 xfun_0.43
[41] tidyselect_1.2.1 rstudioapi_0.16.0 knitr_1.46 htmltools_0.5.8.1
[45] rmarkdown_2.26 compiler_4.4.0